Gegenstand des Projektes ist die konzeptbasierte Strukturierung und semantische Anreicherung der forschungsrelevanten Datenbestände aus klinischen Primärsystemen. Das Ziel ist die Schaffung eines Repositoriums, in dem die klinischen Forschungsdaten der MHH interoperabel, zugänglich und wiederverwendbar sind. Durch den Einsatz semantischer Modellierungstechnologien sind damit klinische Daten für bereichsübergreifende Forschungsvorhaben besser nutzbar.
Um dieses Ziel zu erreichen, müssen die Daten aus den bisher isolierten Systemen herausgelöst und in eine standardisierte, normalisierte und semantisch angereicherte Form gebracht werden. Bisher sind viele identische klinische Konzepte in den jeweiligen Anwendungssystemen unterschiedlich dargestellt, d.h. sie verwenden system- oder fachabteilungsspezifische Datenstrukturen und Vokabularien. Aufgrund dieser mangelnden Standardisierung ist die Bedeutung der Daten oft undurchsichtig, so dass die Durchführung von verlässlichen statistischen Auswertungen sehr viel Arbeitsaufwand in der Datenaufbereitung erfordert. Das treibt die Forschungskosten in die Höhe. Ein anschauliches Beispiel für die hohe Komplexität und Heterogenität sind Beatmungsgeräte, deren unzählige Parameter sich in ihren Bezeichnungen von Hersteller zu Hersteller und in den verschiedenen Anwendungssystemen der MHH erheblich unterscheiden. Um diese Daten für die Auswertung nutzbar zu machen, ist eine Vereinheitlichung von Terminologien und des Datenmodells zwingend notwendig.
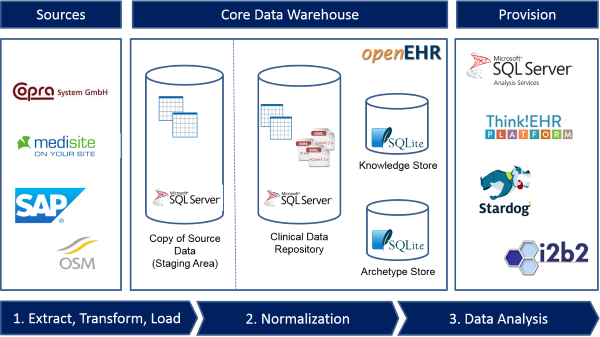
In einem Pilotprojekt wird daher der Einsatz von openEHR Archetypen zur Datenintegration in das Data Warehouse der MHH erprobt. Archetypen sind formalisierte, semantische, maschinenlesbare Modelle klinischer Konzepte, die zwischen Systemen und/oder Menschen ausgetauscht werden können. Ein Archetyp repräsentiert jeweils ein klinisches Konzept (wie z.B. Blutdruck, Blutgasanalyse, Barthel Index oder Apgar-Score) und umfasst sämtliche für die Dokumentation relevanten Datenelemente und Terminologien.
Im ersten Schritt erfolgte die Identifikation der für die Forschung relevanten Daten aus zwei Fachbereichen. Diese umfassen die klinische Dokumentation aus der Intensivmedizin (in den Anwendungssystemen „m.life“ und „COPRA“) und der Chirurgie (in den Anwendungssystemen „i.s.h.med“ und „DoIt“). Der zweite Schritt stellt die detaillierte Analyse der betroffenen Fachbereiche dar, auf deren Grundlage vordefinierte Archetypen ausgewählt oder gänzlich neue erstellt werden. Hierfür ist insbesondere die Einbindung der Forscher, Systemspezialisten, Ärzte und Pflegekräfte aus den unterschiedlichen Fachbereichen wichtig, denn nur in Abstimmung mit ihnen kann garantiert werden, dass die Bedeutung der Daten über die Grenzen der Einrichtungen und Fachdisziplinen hinweg eindeutig verständlich ist. Archetypen helfen bei dieser wichtigen Kommunikation, indem sie die Dokumentation transparent machen und eine für Nicht-Techniker verständliche Darstellung erlauben. Abschließend werden die modellierten Daten in das Data Warehouse überführt.
Auf diese, durch Archetypen beschriebenen Daten kann danach, im Einverständnis mit den Bereichsverantwortlichen sowie unter strenger Wahrung des Datenschutzes, über standardisierte Schnittstellen zugegriffen werden. Dadurch ist es möglich, den Datenimport in spezialisierte Auswertungswerkzeuge zu automatisieren oder Datenbestände direkt in Formate zu überführen, die in klinischen Studien genutzt werden (z.B. CDISC ODM). Folgende Methoden und Werkzeuge sollen eingesetzt werden:
- Semantische Abfragen durch Überführung der Daten in die Web Ontology Language (OWL)
- Analyse von Behandlungsprozessen und Temporal Data Mining mittels ProM
- Kohorten-Identifikation mittels I2B2 und Microsoft Business Intelligence Tools
- Large-Scale Data Mining durch MapReduce-Technologien (z.B. Hadoop)
Der Einsatz von semantischen, maschinenlesbaren Datenformaten zur Datenintegration soll zur Verbesserung der bereichsübergreifenden klinischen Forschung und damit zur Konkurrenzfähigkeit der MHH im Bereich der Wiederverwendung klinischer Datenbestände auf internationalem Niveau beitragen.
Die Arbeiten im Projekt werden in enger Abstimmung mit der Data Warehouse Gruppe des Zentrums für Informationsmanagement (ZIMt) durchgeführt.
Publikationen
- Saalfeld B, Tute E, Wolf KH, Marschollek M. Introducing a Method for Transformation of Paper-Based Research Data into Concept-Based Representation with openEHR. Stud Health Technol Inform. 2017;235:151155.
- Haarbrandt B, Wilschko A, Marschollek M. Modelling of Operative Report Documents for Data Integration into an openEHR-based Enterprise Data Warehouse. Stud Health Technol Inform. 2016;228:407-11.
- Haarbrandt B, Tute E, Marschollek M. Automated Population of an i2b2 Clinical Data Warehouse from an openEHR-based Data Repository. J Biomed Inform. 2016. In Press, Accepted Manuscript
- Tute E. OpenEHR platform concept for semantic interoperability in project HaMSTR. In: Dössel O, editor. Biomedical Engineering Biomedizinische Technik Joint journal of the German Society for Biomedical Engineering in VDE and the Austrian and Swiss Societies for Biomedical Engineering, 2016 · Volume 61 · Issue s1 ISSN 0013-5585. BMT 2016 "Dreiländertagung" Swiss, Austrian and German Societies of Biomedical Engineering; 2016 October 05; Basel, Basel, Swiss. De Gruyter; c2016.
- Haarbrandt B, Marschollek M. Modeling and Integration of Intensive Care Data Into an openEHR-based Enterprise Data Warehouse. Curr Ther Res Clin Exp. 2016;78:Supplement 8-9.
- Haarbrandt B, Jack T, Marschollek M. Automated Transformation of openEHR Data Instances to OWL. Stud Health Technol Inform. 2016;223:63-70.
- Haarbrandt B, Gerbel S, Marschollek M. Einbindung von openEHR Archetypen in den ETL-Prozess eines klinischen Data Warehouse. GMDS 2014. 59. Jahrestagung der Deutschen Gesellschaft für Medizinische Informatik, Biometrie und Epidemiologie e.V. (GMDS). Göttingen, 07.-10.09.2014. Düsseldorf: German Medical Science GMS Publishing House; 2014 (http://www.egms.de/static/de/meetings/gmds2014/14gmds227.shtml)






